
近日,南方科技大学生物医学工程系先进显微成像课题组李依明团队在三维全细胞大视场高通量超分辨成像领域取得重要进展,相关研究成果以 “Field-dependent deep learning enables high-throughput whole-cell 3D super-resolution imaging”为题在国际顶级期刊Nature Methods上长文发表。
具有高对比度和超分辨率的荧光显微镜彻底改变了细胞生物学研究。此外,生物医学研究通常需要大量的统计分析才能得出具有高置信度的结论,具有丰富信息内容的高通量成像被广泛应用于定量生物学研究。一个新兴的趋势是发展用于高内涵筛选的高通量超分辨显微成像技术。基于宽场成像的单分子定位超分辨显微成像技术(single-molecule localization microscopy, SMLM),将光学分辨率提升一个数量级以上,理论上通过使用大面阵探测器就可以在不损失分辨率和速度的条件下,提升成像通量。然而,视场相关的像差通常将SMLM有效的成像区域局限在几十微米的尺度。传统的大视场超分辨成像需要多区域拼接完成,效率非常低下,限制了其对大尺寸样品进行高通量成像。
研究团队提出了一套针对空间移变点扩散函数(point spread function, PSF)精准定位的深度学习算法框架FD-DeepLoc(field dependent deep learning localization)。该框架集成了一套基于GPU加速的矢量PSF全局拟合算法和利用时空信息进行移变单分子定位的深度学习神经网络,在180×180×5 µm3范围内实现了无需扫描的全细胞超分辨成像,将三维SMLM的成像通量提升了约100倍(DOI :10.1038/s41592-023-01775-5)。该工作为超分辨显微成像领域提供了新的技术思路和视角,对研究完整细胞群或组织中纳米级生物结构具有重要的理论意义和应用价值。

为了对大视场中的移变PSF进行精确建模,团队从适用于高数值孔径物镜的矢量PSF建模出发,构建了一套可描述任意位置像差的空间移变PSF模型。结合团队之前发表在Nature Communications的全局拟合算法globLoc对不同z平面的点光源图片进行全局拟合,实现对像差的高精度估计[1]。考虑到建模整个大视场空间移变的像差(21项泽尼克多项式系数表示)需要对整个视场进行上千次的采样,而基于矢量衍射德拜积分理论建立的PSF模型的计算涉及到多次傅里叶变换,计算量非常大。为了加速这一过程,团队开发了领域内第一个基于GPU加速的矢量PSF模型拟合算法和像差校正软件,将校正速度提高了50倍,满足了大视场的PSF建模需求。
近年来,深度学习算法已被广泛应用于科学研究中。凭借出色的特征感知能力和端到端的图像拟合能力,深度学习在SMLM中展现出远超传统算法的性能,尤其是对于分子高度重叠的情况。传统的卷积神经网络对图像中不同的区域使用的是同一个卷积核,对空间位置不敏感。当目标与其空间坐标解耦时,卷积神经网络往往能在目标识别中表现优异。然而,光学像差通常是静态的并且与其位置高度相关。对于大视场成像,需要空间位置敏感的超分辨重建算法。为了克服视场相关的像差,团队在卷积神经网络中引入两个位置相关的通道,卷积核在卷积单分子图片的时候,也对这两个位置通道进行卷积,从而把位置相关的信息编码到神经网络里(图1)。FD-DeepLoc突破了传统卷积神经网络对空间位置不敏感的局限,在全画幅sCMOS相机(1608×1608)视场范围内对空间移变的单分子数据精准定位,实现了三维大视场高通量超分辨成像。
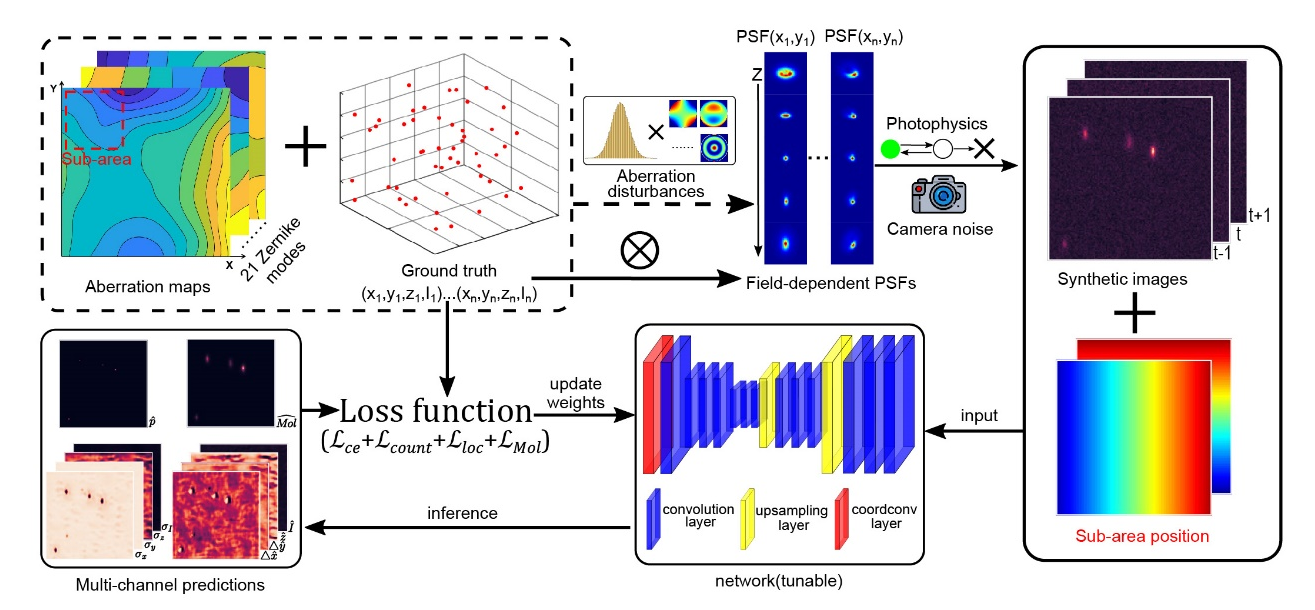
图1 FD-DeepLoc网络框架图
深度学习算法通常需要先验知识对网络进行训练。在FD-DeepLoc网络中,通过精确建模物理成像过程生成海量数据,有效避免了深度学习过拟合的问题。此外,FD-DeepLoc唯一的先验就是PSF模型,不需要预先知道生物样品的结构信息,所以无需对不同的样品重复训练网络。考虑到校准系统PSF时存在测量误差以及样品自身引入的像差,FD-DeepLoc采用鲁棒训练策略,对PSF模型中的像差引入先验分布,同时将不均匀背景建模为柏林噪声,利用上述训练数据得到的网络表现出更好的鲁棒性和精确率。在模拟和实验数据中(图2),FD-DeepLoc相对于目前领域内两个表现最好的算法——团队2018年在Nature Methods上发表的基于三次样条插值的传统单分子拟合Cspline算法[2]和基于神经网络的DECODE算法[3],都有显著的提升。在三维超分辨定位精度指标均方根误差(root-mean-square error, RMSE)上相比于DECODE提升近两倍。此外,FD-DeepLoc包含的大视场空间移变的矢量PSF建模和基于深度学习的单分子定位算法提供开源(https://github.com/Li-Lab-SUSTech/FD-DeepLoc)。
.gif)
图2 FD-DeepLoc用于分析大视场下核孔复合物实验数据
大视场成像可以用来对大尺寸样品进行研究。体外培养神经细胞通常都在几百微米的范围内生长,传统的超分辨成像一般只能看到小于50 µm的轴突片段,往往忽略掉一些不常见的结构,而且无法在更大的尺度上观测神经元之间的精细互作信息。研究人员利用小鼠胚胎干细胞诱导的神经细胞,选用β2血影蛋白作为观测对象。利用传统散光调制的PSF进行三维超分辨成像(图3a-e),血影蛋白沿神经突的周期性组织清晰可见。然而,传统散光PSF的景深有限,难以对哺乳动物5-10μm典型细胞厚度进行全细胞成像,图3d由于重建的超分辨图像景深受限在1 μm左右,离焦处的神经突结构有部分图像丢失。
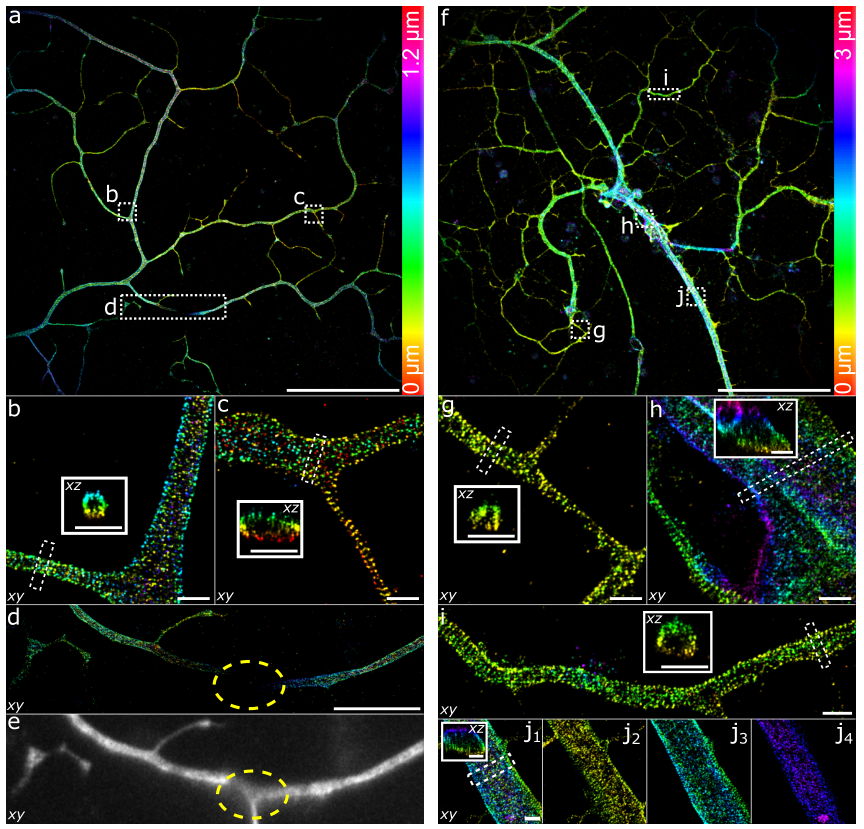
图3 β2血影蛋白在神经细胞中的三维超分辨成像。比例尺,50 μm (a,f),10 μm(d),1 μm(b,c,g–j)
为了拓展轴向成像范围,通常对光瞳函数进行调制得到不同形状的PSF来对不同景深范围的样品进行成像。传统的大景深PSF往往需要复杂的光瞳函数实现,需要用到高像素的空间光调制器。然而,空间光调制器对偏振敏感,用来对荧光信号进行成像会损失一半的光子数。与空间光调制器相反,可变形镜对光子数的利用率比较高,但是能控制的促动器数量较少。为了能精确获得可变形镜调制的光瞳函数,团队提出了一种以可变形镜促动器响应函数为基函数的相位设计框架[4],优化得到了一系列基于可变形镜的三维精度最优PSF(DM-optimized PSF, DMO PSF)。DMO PSF可实现自由景深控制,显著提高了SMLM的图像质量(图4)。对于神经细胞的成像实验,采用可变形镜优化的3µm景深DMO PSF进行数据采集,即使在具有大直径的神经突中,也可以很好地重建血影蛋白的三维分布(图3f-j)。
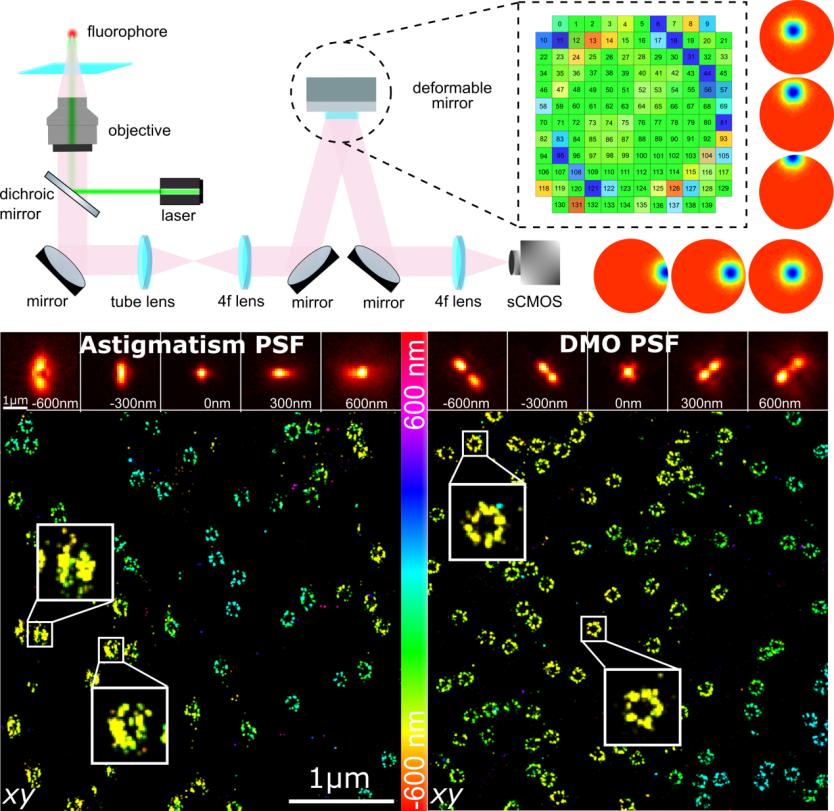
图4 DMO-PSF原理及成像效果
大视场成像也带来了数据通量的提升。得益于高通量成像的能力,研究人员利用 FD-DeepLoc可以在短时间内采集到上百个不经过人工挑选的全细胞线粒体超分辨图像。如图5所示,研究人员在16个感兴趣的视场区域,利用FD-DeepLoc重建出121个全细胞线粒体三维超分辨图像(图5a)。通过提取这些细胞的形态(分叉、长度、球形度等)进行聚类分析,得到三种不同类型的线粒体细胞。如图5b所示,类型1细胞包含更多的小圆形线粒体和更少的分支结构;类型2细胞包含延伸的管状线粒体和更复杂的网络结构;类型3细胞含有球形和管状线粒体的混合物结构。精细的全细胞结构超分辨分析为细胞组学提供了新思路。
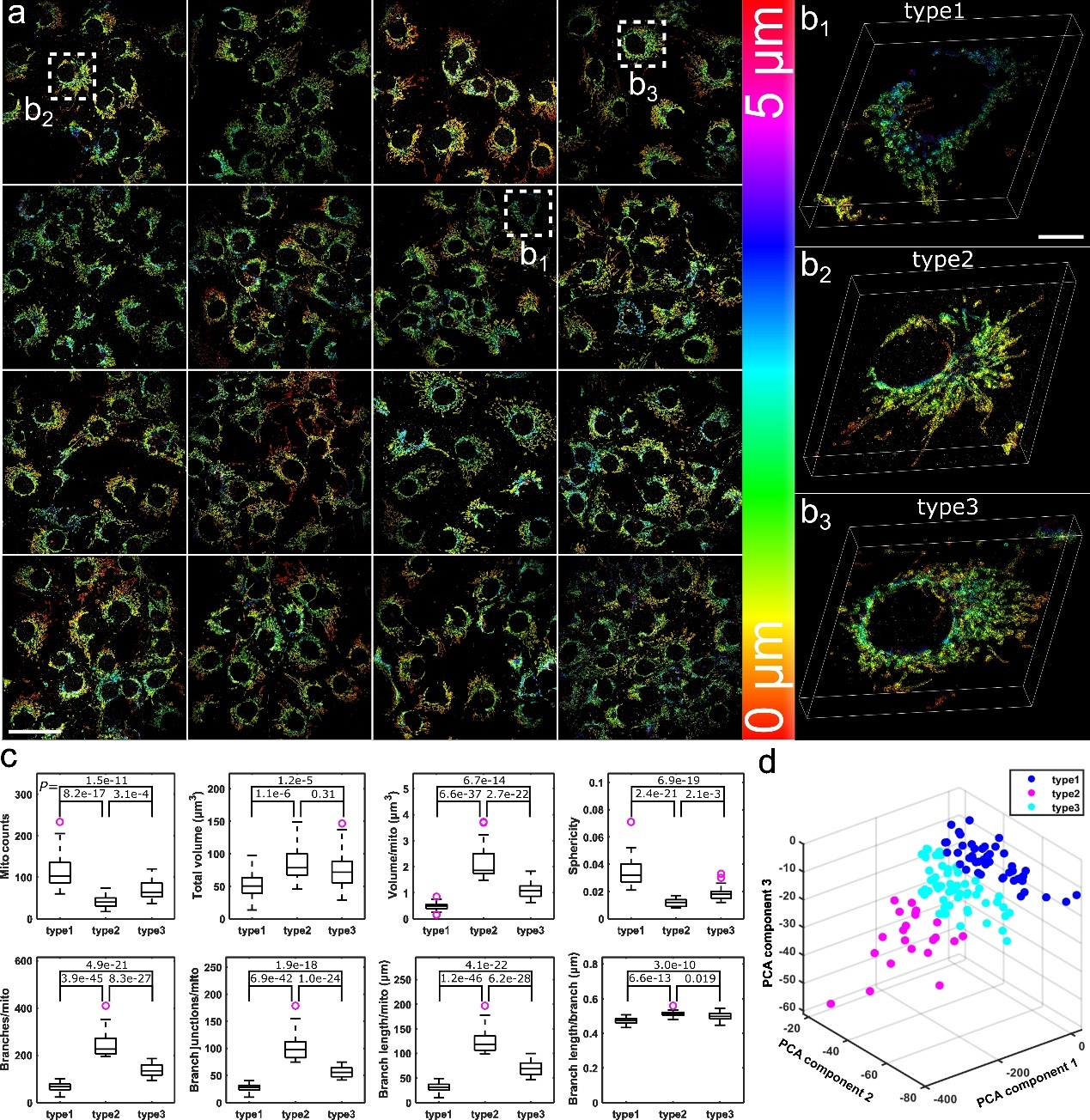
图5 全细胞三维超分辨线粒体图像的定量分析。比例尺,50 μm(a),10 μm(b1)
综上所述,该团队提出了一种可以对空间移变PSF进行精确定位的深度学习算法,结合GPU加速的矢量PSF拟合算法对大视场像差进行快速精准建模和基于可变形镜调制的DMO PSF自由景深控制,实现了三维全细胞大视场高通量超分辨成像。相比于传统基于散光的三维SMLM成像,FD-DeepLoc将SMLM的成像通量提高了约100倍,充分利用sCMOS的全画幅面阵实现高保真的生物结构重建和高精度体积成像。该工作无需复杂的硬件自动化即可将SMLM从低通量技术转变为高通量成像技术,使大尺寸样品的体积三维超分辨成像成为可能,这有助于生物学研究中罕见结构或事件的发现,并提高生物学研究中的定量统计分析能力。未来可利用最新更大面阵的sCMOS相机,对进一步提升超分辨成像的通量至毫米级尺寸,可为脑切片、病理样品和类器官等大尺寸样品成像提供新的技术手段,使研究人员在大尺寸厚样品原位解析生物大分子结构的同时分析细胞种群间的微小差异,为结构生物学、细胞生物学的研究提供新视角。
南方科技大学生物医学工程系的李依明副教授为该论文的通讯作者,在读博士生傅爽和石伟为论文的共同一作,南方科技大学为第一通讯单位。该项目得到了山东省重点研发计划,深圳市孔雀团队和南方科技大学启动资金等科研项目的支持。
原文链接:https://www.nature.com/articles/s41592-023-01775-5
参考文献:
1. Li, Y. et al. Global fitting for high-accuracy multi-channel single-molecule localization. Nat. Commun. 13, 3133 (2022).
2.Li, Y. et al. Real-time 3D single-molecule localization using experimental point spread functions. Nat. Methods 15, 367–369 (2018).
3.Speiser, A. et al. Deep learning enables fast and dense single-molecule localization with high accuracy. Nat. Methods 18, 1082–1090 (2021).
4.Fu, S. et al. Deformable mirror based optimal PSF engineering for 3D super-resolution imaging. Opt. Lett. 47, 3031 (2022).
撰文:石伟,傅爽
审核修改:李依明
作者简介:
李依明,南方科技大学研究员,2009、2010、2015年分别于上海交通大学、海德堡大学、卡尔斯鲁厄理工学院获得生物医学工程学士、医学物理硕士和生物物理博士学位。2016-2019年受玛丽居里博士后奖学金资助,分别在欧洲分子生物实验室和耶鲁大学任职博士后和访问学者。长期致力于研究三维超高分辨显微成像技术及其生物应用,在光学仪器研发,光学理论和成像算法等方面均有一定的积累,近五年来发表高影响力论文13篇,其中第一/通讯作者论文7篇,包括Nature Methods(2篇),Nature Communications(1篇),Optics Letters(3篇)等。入选“深圳市海外高层次人才B类”,目前和包括来自EMBL,耶鲁大学,牛津大学和剑桥大学等多个实验室有合作关系。课题组长期招收博士后、研究助理及博士/硕士研究生,有物理,光学工程、计算机、机械工程、生物医学工程、生物技术、生物化学等相关专业背景,要求具备良好的学习能力、独立工作能力和团队沟通能力。
课题组主页:https://li-lab-sustech.github.io/
有意者请将个人简历发送至李依明副教授邮箱:liym2019@sustech.edu.cn。

研究团队人员合影(通讯作者前排中间,共同一作前排左4和前排右1)