近日,南方科技大学生物医学工程系李喆副教授和普渡大学DNA纳米技术专家毛成德教授在学术期刊Science上发表了题为“Engineering colloidal crystals molecule by molecule”的展望性评述文章[1](图1)。该评述介绍了同期Science期刊发表的两篇用DNA分子精确可控构建胶体晶体的研究论文,并对工作中的研究背景、技术要点和发展前景进行了评述。
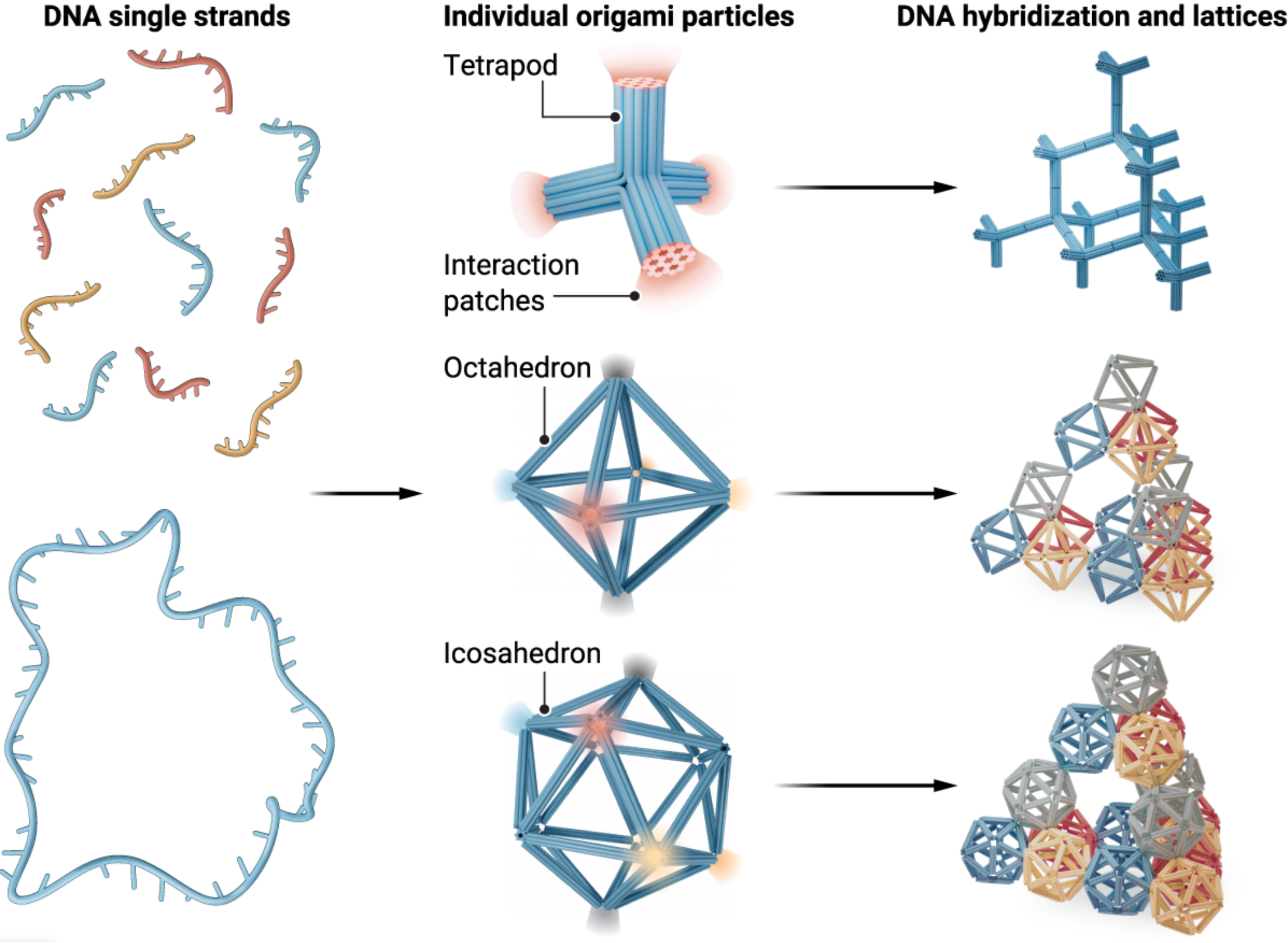
图1 DNA胶体晶体的工程化构建[1]
基于分子设计的晶体工程已经为医学、催化、光学和电子学等领域带来了许多科学技术进步。区别于分子晶体,胶体晶体通常是由胶体粒子而非单个分子排列而成的高度有序结构。虽然科学家们对胶体晶体进行了长期的研究,但是与分子晶体所能达到的控制水平相比,在胶体晶体中实现高精度的结构设计和复杂的编程调控仍然是一个重要的挑战。
传统的胶体粒子在尺寸均匀性、形状控制、多分散性以及颗粒间相互作用的调节上存在许多局限性。相比之下,DNA 折纸——一种由成百上千条DNA单链自组装形成的纳米结构,可以对这些性质进行纳米级别精度的编程。尽管十多年前人们就意识到了 DNA 折纸在胶体组装中的潜力,但是实现DNA胶体晶体经历了漫长的试验和犯错。编程DNA胶体晶体存在许多挑战:例如,DNA结构和相互作用的柔性可能导致组装坍塌或扭曲;非特异性相互作用可能导致错误组装;过强的相互作用设计可能让DNA折纸微粒进入动力学陷阱而发生无规聚集,阻碍它们组装成热力学上更稳定的有序结构。
在最新一期Science中,慕尼黑大学Tim Liedl课题组Gregor Posnjak等人创建了一种基于DNA折纸的立方金刚石胶体晶体[2]。这种胶体晶体中,DNA折纸颗粒以与金刚石中碳原子相同的方式排列。金刚石这类晶格具有大的开放空间,从能量的角度来说是不稳定的,因此在过去的胶体晶体设计中的成功率很低。作者设计了一种DNA折纸四足体来实现金刚石胶体晶体的组装。四足体的四个臂沿着正四面体的方向延伸,每个臂的末端是单链DNA尾部。尾部携带互补的DNA序列,能够相互杂交引导相邻的四足体以60°交错的扭转角度连接。研究发现,两个相邻的四足体间需要形成交错而非重叠的构象才能引导正确的晶体组装。同时,DNA杂交的相互作用强度必须足够微弱,才能组装出晶面清晰、有序性强的金刚石胶体晶体。

图2 DNA折纸金刚石胶体晶体的分子设计和自组装[2]
在同期Science的另一篇研究工作中,亚利桑那州立大学Petr Šulc课题组Hao Liu等人建立了一个强大的计算实验框架,构建基于 DNA 折纸的胶体晶体自组装[3]。在模拟中,作者将 DNA 折纸视为表面上具有多个相互作用区域的球形颗粒,并使用反馈回路来推进有利于晶格形成的设计,并排除导致竞争性组装的设计。经过一定次数的迭代,作者在计算机上高产率地获得了烧绿石晶格(一种正四面体顶点相互连接的三维排列)。作者进一步在实验中将DNA八面体以及二十面体折纸组装成烧绿石晶格,证实了模拟结果。
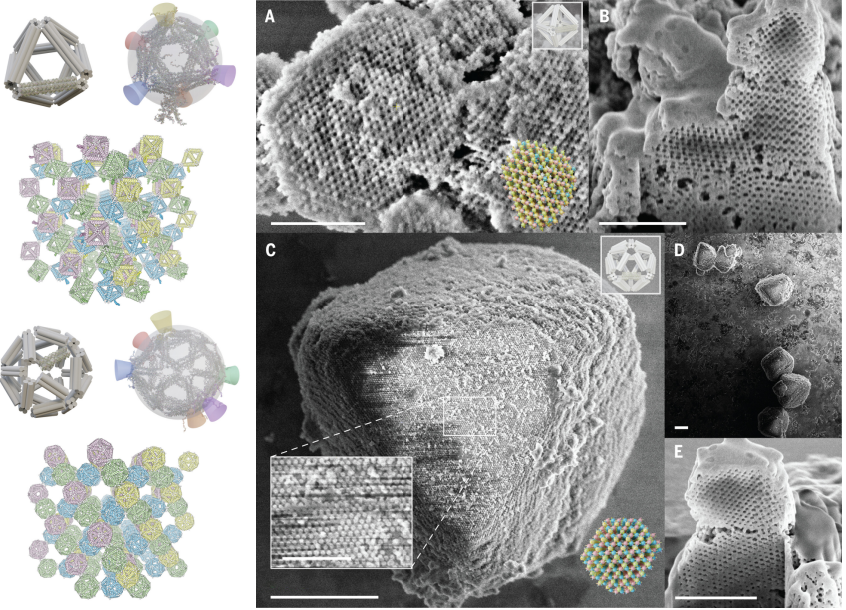
图3 DNA折纸烧绿石胶体晶体的分子设计和自组装[3]
在评述中,李喆副教授和毛诚德教授指出,成功组装DNA胶体晶体的关键在于颗粒间相互作用的设计。在Posnjak等人的方法中,相邻四足体之间多个分子相互作用可以约束四足体的交错角度促进晶体组装;在Petr Šulc等人的方法中,模拟方法搜索和确定了多组相互作用,利用多价性和特异性将DNA折纸颗粒固定并堆积在所需的晶格位置。评述最后还对DNA胶体晶体领域的发展进行了展望:进一步调整设计参数和实验条件有望实现尺寸更大、组装更为有序的DNA胶体晶体,这会影响胶体晶体的性质并进步拓展它们的应用。通过设计单个DNA折纸颗粒及其相互作用,有望在胶体晶体实现前所未有的复杂晶体结构。这些新型的胶体晶体可以在与紫外线和可见光波长相当的长度尺度上排列材料元件,构建光学超材料。结合动态DNA纳米技术,这些高度可编程的胶体晶体可以被进一步设计成响应性和可重构的光学器件,并能够以以前无法达到的精度来操纵光。
南方科技大学为论文的第一单位,生物医学工程系李喆副教授是评述论文的第一作者和共同通讯作者。李喆副教授多年来致力于生物纳米技术的研究,特别是生物大分子的晶体设计。他在博士和博士后期间分别师从DNA纳米技术领域专家毛成德教授和蛋白质设计专家、美国科学院院士David Baker教授,提出了多种原创性的晶体设计思路和改造方法,包括高稳定性和动态响应性DNA三维晶体设计[4,5],以及蛋白质三维晶体的计算设计[6]。例如,在2023年Nature Materials期刊上题为“Accurate computational design of three-dimensional protein crystals”的论文中, 李喆博士、王顺智博士和Una Nattermann博士提出了一种精确设计蛋白质三维晶体的通用方法。这种开创性的方法实现了原子级别精确的蛋白质晶体设计,并且在蛋白质分子的初级序列中编码了晶体材料的自组装信息,为结构生物学研究和生物材料工程提供了强大的平台。
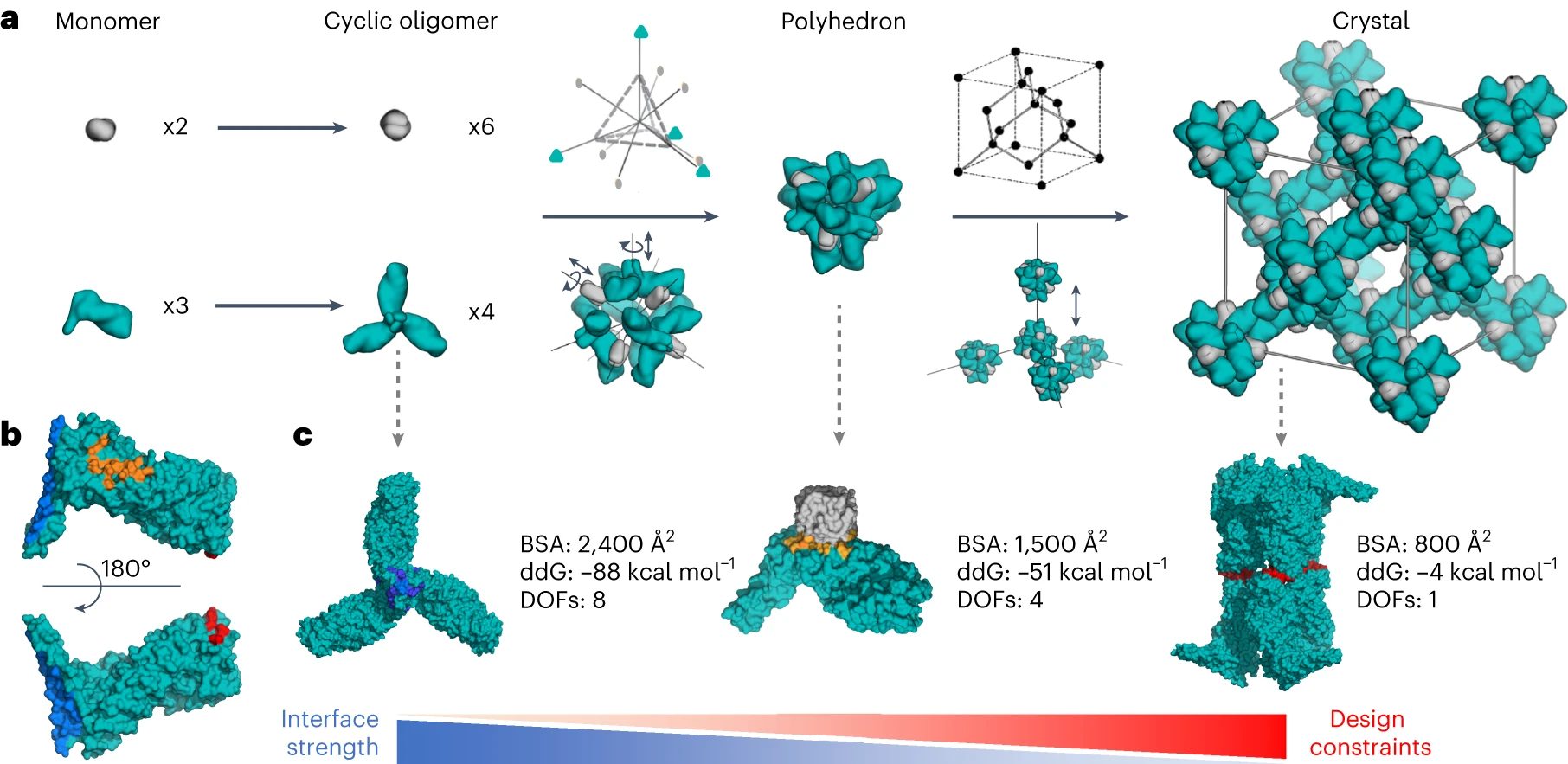
图4 蛋白质三维晶体的计算设计[6]
论文链接:
[1] https://www.science.org/doi/10.1126/science.adp4370
[2] https://www.science.org/doi/10.1126/science.adl2733
[3] https://www.science.org/doi/10.1126/science.adl5549
[4] https://pubs.acs.org/doi/10.1021/jacs.9b06613
[5] https://onlinelibrary.wiley.com/doi/10.1002/adma.202200441?af=R